赵方庆团队提出面向TB级数据的环形RNA高效挖掘算法
环形RNA因其独特的共价闭合环状结构,能够有效抵抗细胞内核酸外切酶的降解,展现出远高于线性RNA的稳定性,不仅成为基因调控与疾病机制研究的前沿热点,也作为多种重大疾病的生物标志物与潜在治疗靶点而备受关注。然而,现有环形RNA识别与定量算法流程复杂、资源消耗巨大,且在识别精度和灵敏度方面仍存在不足,难以实现大规模数据中低丰度及非典型环形RNA的高效检测,极大制约了基于现有海量转录组测序数据集的环形RNA研究的系统开展。
2025年10月1日,中国科学院动物研究所赵方庆团队与国家生物信息中心高远团队合作在Nature Biotechnology发表了题为Detecting and quantifying circular RNAs in terabyte-scale RNA-seq datasets with CIRI3的研究论文。该研究提出了面向TB级转录组数据的高效环形RNA检测与定量工具CIRI3,通过反向剪接序列比对与跨样本整合的创新算法设计,实现了对TB级数据的超高速处理。CIRI3算法不仅显著提升了低丰度环形RNA的检出能力,还拓展了对非典型剪接形式的识别覆盖,有效突破了当前环形RNA大规模检测在灵敏度与计算效率方面的技术瓶颈。
针对现有环形RNA 计算工具效率低、内存占用大的问题,CIRI3创新性地采用动态多线程任务划分与分块查找策略,实现了TB级测序数据的超高效处理。相较于单一样本分析,基于CIRI3的大样本联合分析可有效防止“漏检”,使得环形RNA检出率增加近20%,并显著提升了低丰度环形RNA的检测灵敏度与定量精度。此外,CIRI3对非经典剪接信号等关键特征具有高度的可扩展性,首次实现了对内含子自连接型环状RNA等新亚型的系统性识别。综合性能评估表明,CIRI3在环形RNA的识别与定量方面均显著优于现有工具,为大规模环形RNA研究及疾病生物标志物的系统性挖掘提供了关键的技术支撑。
基于CIRI3的大规模数据挖掘能力,研究团队对涵盖30种癌症类型的2,535个人类癌症及正常样本转录组测序数据进行了系统整合挖掘,构建了迄今规模最大的AI-ready肿瘤相关环形RNA数据资源CIRIonco。进一步评估表明,CIRI3计算的环形RNA剪接比率在不同批次和不同来源的样本间表现出高度的稳健性,其批次效应显著低于传统的反向剪接读段数,使其成为跨批次、跨平台整合分析的更优指标。研究团队进一步构建了基于人工智能的环形RNA溯源与疾病表型预测模型,实现了样本来源器官系统、组织类型及疾病水平的精准预测。该模型还可在用户小规模数据集上进行迁移学习,实现优于从头训练模型的预测性能,为解析环形RNA的生成调控与功能机制提供了重要的计算工具。
综上所述,CIRI3是一个可在TB级人群队列转录组数据中高效检测和精确定量环形RNA的工具,同时CIRIonco数据库为癌症相关环形RNA的研究与功能探索提供了重要的AI-ready数据集与计算模型,为环形RNA在癌症分型和精准诊断中的应用提供了方法学工具和数据基础。
国家生物信息中心高远研究员与中国科学院动物研究所赵方庆研究员为该研究的通讯作者,国家生物信息中心博士研究生郑鑫、动物研究所张金阳副研究员及国家生物信息中心宋利璞助理研究员为共同第一作者。该研究获得国家重点研发计划、中国科学院战略性先导科技专项、国家自然科学基金、北京市自然科学基金及中国科学院人才计划等资助。
文章链接:https://doi.org/10.1038/s41587-025-02835-1
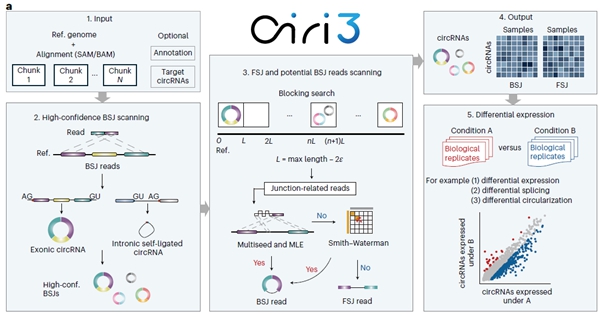
图1. CIRI3算法原理

在演化生物学中,趋同演化是指不同生物类群在相似环境压力下独立演化出同一性状的现象。为了挖掘这种适应性演化的分子基础,目前的比较基因组学研究多关注于氨基酸“状态趋同”,即不同物种在相同的蛋白序列位点...

趋同演化是生物演化中的重要现象,指不同类群的生物在适应相似环境的过程中,独立演化出相似的功能性状,例如鸟类、蝙蝠、翼龙独立演化出扑翼飞行能力。

