张勇团队合作开发低DNA用量、无扩增的PacBio建库技术LILAP
基因组信息在生命科学研究中具有重要价值。随着价格的下降,基于三代长读长的测序组装越来越广泛地应用于基因组学研究(Nat Methods 2022;Nat Rev Gene 2024);其中,PacBio高保真(High-fidelity,HiFi)测序技术因其高精度的测序质量被认为是三代测序的金标准。然而,其标准建库方法需要大量DNA输入(>1 μg),限制了其在小型昆虫或珍稀样本中的应用(Proc Natl Acad Sci U S A 2021; Nucleic Acids Res 2020)。DNA扩增虽然可以降低样本输入量,但会引入扩增偏好和人工嵌合序列等错误,影响基因组的完整性及变异检测的准确性。因此,低DNA用量、无扩增且操作简单的PacBio建库方法亟待开发。
2024年7月5日,中国科学院动物研究所张勇研究组与中国农业科学院深圳农业基因组研究所阮珏研究组在Nature Communications杂志上在线发表了题为“Low-input PacBio sequencing generates high-quality individual fly genomes and characterizes mutational processes”的论文。该研究开发了一种基于Tn5转座酶的低DNA用量(100 ng)、无扩增、低成本的PacBio建库技术LILAP。团队利用该技术实现了两个单只雄性黑腹果蝇的全基因组测序和高质量组装,同时获得了内共生菌Wolbachia完整基因组,展示出LILAP的应用价值。基因组分析还发现了两个突变特性:复杂转座事件更倾向于发生在non-B DNA序列富集的区域;基因转换(gene conversion)事件使转座子同质化。
首先,LILAP利用Tn5转座酶和发卡结构的PacBio测序接头组成二聚体转座复合物,一步实现DNA片段化和测序接头连接(图1)。所有流程在单个试管内进行,最大限度减少了建库过程中的DNA损失,简化了建库流程。与PacBio公司的标准建库和低DNA扩增建库方法不同(Genes 2019;Genome Biol 2021),LILAP无需特殊实验仪器,所有试剂均可在第三方公司购买,建库成本降至每个样品10美元。同时,测序接头内还可添加条形码(barcode),用于多样本混合建库测序(Genome Biol 2010)。
研究人员进一步使用黑腹果蝇ISO1品系对该技术进行评估,结果显示两个单只果蝇的读段测序质量中位数达到36(即碱基错误率低于0.02%)。从头组装的基因组N50数值超过6 MB,质量分数(QV)超过60(即碱基错误率低于10-6),保守单拷贝基因(BUSCO)组装覆盖度大于98%,远超已发表工作中单只果蝇基因组的质量。
此外,研究者从基因组组装结果中检测到337个新近产生的结构变异,其中9个复杂结构变异为转座子插入引发序列重复或缺失。这些复杂事件涉及两种转座子:DNA转座子hobo (EMBO J 1986)和LTR逆转座子Jockey (Microbiol Spectr 2015)。研究还发现复杂结构变异插入位点附近富集non-B DNA序列(Trends Genet 2023),并提出转座插入后继续发生双链断裂,随后易于出错的DNA修复过程发生。
类似于结构变异,三代长读长测序也使成簇的串联单核苷酸多态性(clustered SNP,cSNP)的检测更为精准。两个单只果蝇总检测到1756个SNP位点,54.9%彼此临近,位于1000 bp窗口内。32.5%的cSNP由转座子贡献,在基因组内可找到供体,表明基因转换贡献了cSNP突变产生 (Nature 2023)。进一步的分析结果支持基因转换可在DNA和RNA水平同时发生。
最后,研究人员在全基因组组装结果中发现了内共生微生物Wolbachia pipientis完整基因组,该微生物广泛分布于节肢动物中。研究团队检测了Wolbachia基因组内变异情况,发现了3个新突变,为后续研究共生菌和宿主的互作提供了新的线索。
综上所述,LILAP作为一种三代低DNA输入量的建库方法,在不扩增的前提下将PacBio测序DNA投入量降低至100 ng,适应于解析小型生物及其内共生体基因组。未来,LILAP可应用于更多场景(图2): DNA用量受限的小型生物多样性探究及较大物种的体细胞变异分析;果蝇个体水平的全基因组关联分析(GWAS);宿主-共生体的协同进化研究。
张勇研究组聚焦重复序列的突变机制、功能演化及转化研究,在重复基因与转座子两方面有较深的积累 (Cell 2024;Nature Ecology & Evolution 2022/2024;Nature Biotechnology 2023)。为精准解析重复序列,研究组近年来致力于测序技术的开发;LILAP与已发表的二、三代高精度测序技术综述(Genomics,Proteomics & Bioinformatics 2024)为该方向的近期进展。LILAP研究的第一作者为中国科学院动物所博士后贾行星、副研究员谭生军、博士生蔡英傲。通讯作者除张勇、阮珏外还包括贾行星与谭生军。郭言言、沈洁宇、张雅琼、马慧静、张青竹、陈金锋、乔格侠等合作者在文章写作、实验和计算分析等方面提供了大力支持。该研究得到了国家重点研发计划(2019YFA0802600),国家自然科学基金(32325014)和中国科学院基础前沿科学研究计划(ZDBS-LY-SM005)的支持。
论文链接: https://www.nature.com/articles/s41467-024-49992-6
代表性参考文献:
Mao Y,Zhang G. A complete,telomere-to-telomere human genome sequence presents new opportunities for evolutionary genomics. Nat Methods 19,635-638 (2022).
Li H,Durbin R. Genome assembly in the telomere-to-telomere era. Nat Rev Genet (2024).
Hotaling S,Kelley JL,Frandsen PB. Toward a genome sequence for every animal: Where are we now?Proc Natl Acad Sci U S A 118,e2109019118 (2021).
Adams M,et al. One fly-one genome: chromosome-scale genome assembly of a single outbred Drosophila melanogaster. Nucleic Acids Res 48,e75 (2020).
Kingan SB,et al. A high-quality de novo genome assembly from a single mosquito using PacBio sequencing. Genes 10,62 (2019).
Fan X,et al. SMOOTH-seq: single-cell genome sequencing of human cells on a third-generation sequencing platform. Genome Biol 22,195 (2021).
Adey A,et al. Rapid,low-input,low-bias construction of shotgun fragment libraries by high-density in vitro transposition. Genome Biol 11,R119 (2010).
Streck RD,Macgaffey JE,Beckendorf SK. The structure of hobo transposable elements and their insertion sites. EMBO J 5,3615-3623 (1986).
Richardson SR,Doucet AJ,Kopera HC,Moldovan JB,Garcia-Perez JL,Moran JV. The Influence of LINE-1 and SINE Retrotransposons on Mammalian Genomes. Microbiol Spectr 3,MDNA3-0061-2014 (2015).
Makova KD,Weissensteiner MH. Noncanonical DNA structures are drivers of genome evolution. Trends Genet 39,109-124 (2023).
Vollger MR,et al. Increased mutation and gene conversion within human segmental duplications.Nature 617,325-334 (2023).
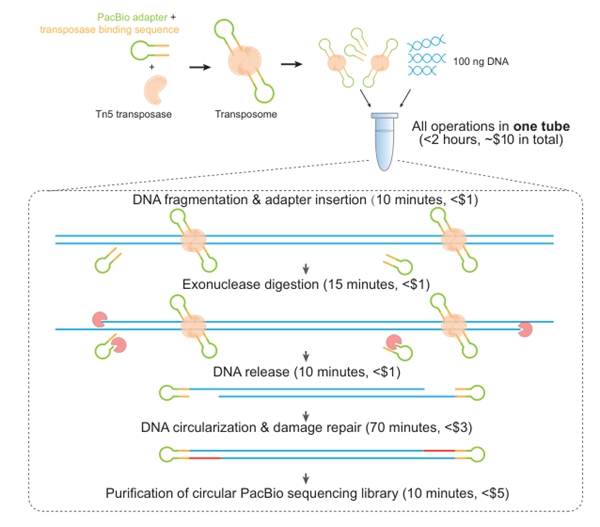
图1. LILAP测序技术原理示意图
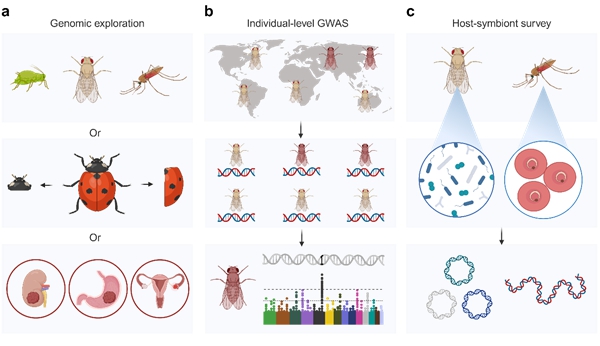
图2. LILAP的潜在应用场景。a) 多样性基因组学及体细胞突变检测。b) 果蝇个体水平的GWAS研究。c) 宿主-共生体解析。
替代性繁殖策略(Alternative reproductive tactics,ART)是指同一性别的个体由于竞争而进化出来的不同的繁殖策略,可以理解为:动物界中,为了繁殖成功这个目的,同时又为了避免主流竞争和对抗,而采取“剑走偏...

外来物种入侵正导致全球生物多样性的快速下降,对外来物种进行早期预警对防治外来物种侵害至为关键。外来物种入侵数量与环境容纳量密切相关,揭示外来物种数量快速增长的栖息地面积阈值,对于预测外来物种入侵防...
为了探索黄胸鼠低温耐受能力和进一步北扩的可能范围,尤其能否继续扩张到东北地区,与褐家鼠东北种群重叠而形成新的危害,研究人员对全国范围内的褐家鼠和黄胸鼠样本进行了全基因组分析,并对室内群体进行低温驯...

